import os, glob
import json
import pandas as pd
from tqdm.auto import tqdm
tqdm.pandas()
# 폴더 안에 파일들을 각각 읽기 위함
DATA_PATH = './kor_ner_data'
folders = os.listdir(DATA_PATH)
folder_1_path = os.path.join(DATA_PATH, folders[0])
folder_2_path = os.path.join(DATA_PATH, folders[1])
folder_1_file_list = os.listdir(folder_1_path)
folder_2_file_list = os.listdir(folder_2_path)
# 확실히 800MB가 넘는 파일들을 다루다보니 주피터가 테스트 하던중 잘 터짐.. 이유는
# python pandas 는 in-memory 처리 방식 이기 때문인듯
# 이번에 간당간당 하게 넘어갔지만 다음에는 파이스파크 써봐야 할것같다..
# https://3months.tistory.com/566
# pyspark 환경에서는 메모리 사용량을 최소화하는 방식으로 용량이 크고,
# 포맷이 다양한 데이터들을 "특정 데이터 구조" 로 로드하고 처리하는 것이 가능하다.
# 즉, pyspark 는 시간 및 컴퓨팅 자원 측면에서 효율적으로 데이터 처리/분석을 할 수 있도록 도와준다.
file_path = os.path.join(folder_1_path, folder_1_file_list[0]) # 이 폴더는 파일이 하나였음
df = pd.read_json(file_path, orient='index').T
df['metadata'][0]


counter = 0
tmp = []
for datum in tqdm(df['document'][0]):
for idx, sentences in enumerate(datum['sentence']):
for sentence in sentences['NE']:
tmp.append((sentence['label'], sentence['form']))
# 여기는 폴더안에 json 파일이 270개 있음
df_2 = pd.DataFrame()
for file_name in folder_2_file_list:
file_path = os.path.join(folder_2_path, file_name)
df_2 = pd.concat((df_2, pd.read_json(file_path, orient='index').T), axis = 0)위와 마찬가지로 NE : named entity만 추출할것임

tmp2 = []
for i, datum in df_2[:].iterrows():
for d in datum['document'][0]['sentence']:
for idx, sentence in enumerate(d['NE']):
tmp2.append((sentence['label'], sentence['form']))추출한 테그들의 중복을 제거하면 중복되지 않는 82527개의 개체들이 태깅된것을 확인할 수 있음

개체명 사전을 하나로 묶고 각각의 개체들을 리스트 형태로 담아서 사전형으로 변환
# https://www.geeksforgeeks.org/python-convert-a-list-of-tuples-into-dictionary/
def Convert(tup, di):
for a, b in tup:
di.setdefault(a, []).append(b)
return di
ne_tag_set_accumulate = {}
named_entities_set = Convert(ne_tag_set, ne_tag_set_accumulate)데이터 프레임으로 변환

최종 추출 결과: 이 개체들을 사전으로 활용하여 문장 augmentation에 사용해볼수도 있겠다.


해당 컬럼들은 _를 기준으로 소분류로 세세하게 테그가 되어있다. TMI_EMAIL태그는 3개 밖에 없었다.
TM에 속한 태그들은 TM, TMM, TMI 로 세세하게 날씨, 의학, 인터넷 등 각종 정보별로 분류가 되어있었다.
데이터셋은 불균형하지만 cross entropy loss를 사용하여 개체명 인식모델이 학습되기때문에 각 태그별 성능에는 서로 영향은 없을것으로 보이지만, 확실히 이메일 처럼 3개 밖에 없는 경우에는 학습셋에 전부 넣지 않는이상 잘 잡지 못할것으로 보인다. 이런것들은 데이터 증강이 필요해보임
경험상 개체명 인식은 중의성 해소를 위해 도메인별로 분리하는게 좋다.
예를들어 책 이름을 인식하는 개체가 들어간 인식모델을 만든다고 가정할때, BOK (책) 개체가 있고, EVT (사건) 라는 개체가 있는데, 책 제목이 하필 '철교 살인 사건'처럼 중의성이 해소되지 않는다면 올바른 정보를 추출하기 어려울 수도 있다.
다음처럼 세분류들을 대분류(main category)로 묶어보았다.
def Convert2main_category(tup, di):
for a, b in tup:
a = a.split('_')[0]
if 'TM' in a:
a = 'TM'
if 'LC' in a:
a = 'LC'
if 'AF' in a:
a = 'AF'
di.setdefault(a, []).append(b)
return di
main_ne_tag_set_accumulate = {}
main_named_entities_set = Convert2main_category(ne_tag_set[:], main_ne_tag_set_accumulate)
대분류 개체들을 막대차트로 나타내보았다.

다음은 2000개 미만으로 보이는 태그 : MT, AM, FD, TI, PT에 대해서 일부만 살펴본 결과이다.
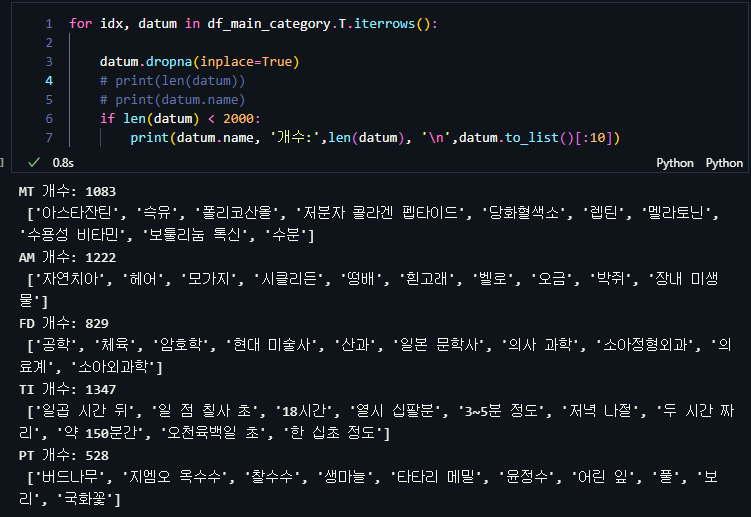
MT: material 과 같은 정보는 확실히 과학 도메인에서 다루면 좋지 않을까
AM : 동물
FD : study field(전문 분야) 도 각 분야별로 개체명 인식모델이 생성되면 더 정확해 보인다.
TI : 시간은 간단한 규칙으로 증강이 가능해 보인다.
PT : plant(식물) 에 윤정수가 들어있는건 피할수 없는 human error이다.
범용적 개체명 인식모델로 대분류적인 개체들을 인식하는 모델을 만들고,
범용적 개체명 인식모델을 통해 특정 도메인에서 범용적인 개체들을 인식 후, 추가적인 세부 개체들을 만들어 나가는 방법이 향후 도메인별 개체명 인식모델의 개발에 효율적일 것으로 생각된다.
참고하기 좋은 논문 (NER)
https://koreascience.kr/article/JAKO202209542016700.pdf
'Machine Learning > DL - NLP' 카테고리의 다른 글
| Chroma(Vector DB) and Sentence Transformer (0) | 2023.05.30 |
|---|---|
| LLaMA 모델의 간략한 역사 (0) | 2023.05.23 |
| NLP모델 파라미터 수 알아보기(feat. number of parameters of DNN models) (0) | 2023.01.12 |
| 사전학습 언어모델 추가학습 (feat. KoElectra, GCP) (1) | 2022.10.03 |
| Got me looking for attention (feat. Self-attention) (0) | 2022.09.19 |


댓글